Results for WGCNA 'female liver' tutorial data set, having 3600 probes and 135 samples
Sage Coexpression, organic correlation algorithm |
Sage Coexpression, WGCNA correlation algorithm |
WGCNA Coexpression |
|---|---|---|
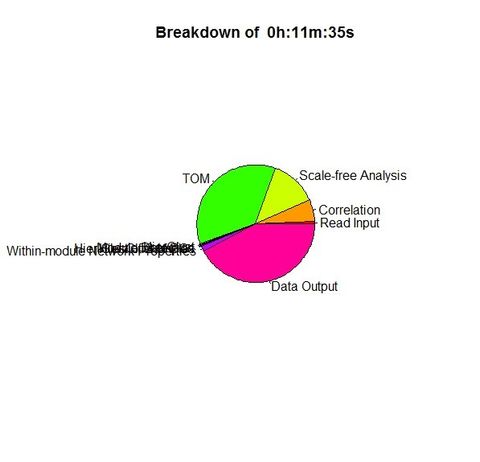
Unix space ('M' field from /usr/bin/time): 6471520KB (or 6.47GB) |
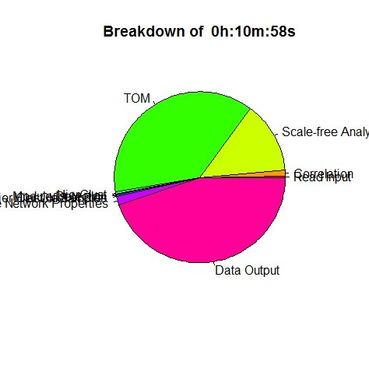
Unix space ('M' field from /usr/bin/time):: 6474832K (6.47GB) |
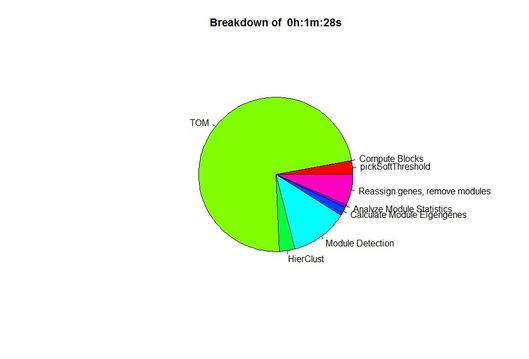
Unix space ('M' field from /usr/bin/time):: 1958480KB (1.96GB) |
Unix time('E' field from /usr/bin/time): 14:04.38elapsed |
Unix time('E' field from /usr/bin/time):13:33.74elapsed |
Unix time('E' field from /usr/bin/time):2:07.23elapsed |
Unix space #2: 6472128 (6.47GB) |
Unix space #2: 6090192 (6.1GB) |
Unix space #2: 1922800 (1.9GB) |
Unix time #2: 11:36.09elapsed |
Unix time #2: 10:58.03elapsed |
Unix time #2: 1:28.53elapsed |
|
|
|
Module difference vs. this baseline (% of pairs for which the algorithm disagrees with this baseline on module co-membership): |
0 |
7.4% |
Module co-membership comparison (vs. this column as a baseline): |
turquoise blue brown yellow green red grey black pink magenta purple greenyellow tan salmon cyan midnightblue lightcyan |
yellow brown blue green turquoise red black pink magenta grey greenyellow purple tan salmon cyan lightcyan midnightblue |
|
<<< Sage dendrogram and modules, with WGCNA modules underneath, for comparison. |
|
TOM Comparison
We captured the TOM matrices from the Sage and WGCNA algorithms. The values are the same (except for some small differences which look like rounding/accuracy differences) as illustrated in this scatter plot:
Note: To make the TOM values the same for both algorithms, the default value for the WGCNA parameter "tomType" is overridden, changing it from "signed" to "unsigned". The definition of the parameter is given here: http://www.genetics.ucla.edu/labs/horvath/CoexpressionNetwork/Rpackages/WGCNA/OldPackages/WGCNA_0.67-2.pdf
- TOMType: a character string specifying TOM type to be calculated. One of "unsigned", "signed". If "unsigned", the standard TOM will be used (more generally, TOM function will receive the adjacency as input). If "signed", TOM will keep track of the sign of the adjacency between neighbors. ... If TOMType = "unsigned", entries of the adjacency matrix are required to lie between 0 and 1; for TOMType = "signed" they can be between -1 and 1.
The TOM values are the inputs to the clustering algorithms. The dendrograms appear nearly identical above, therefore it seems the differences between the two algorithms are in the module determination from the dendrograms.



0 Comments